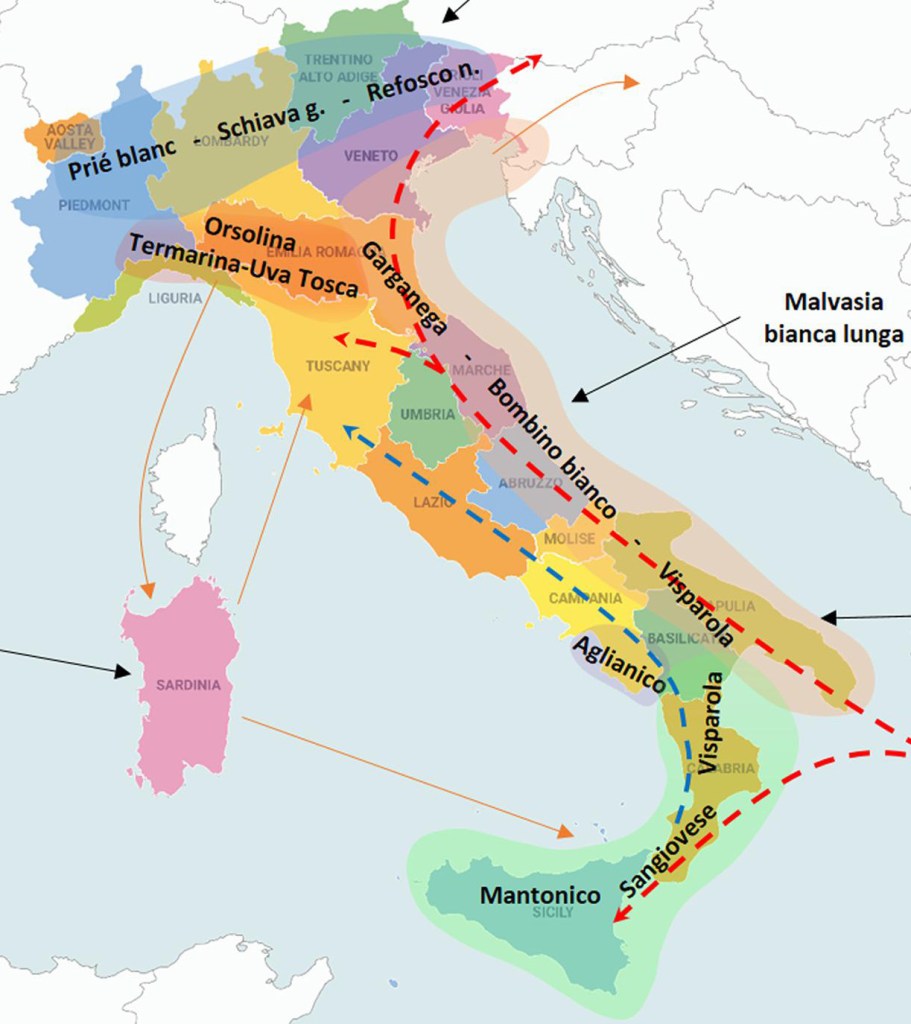
Centralità della “Visparola” nell’origine del germoplasma viticolo italiano, pianta che nei secoli è letteralmente migrata da Sud a Nord: con apparentamenti successivi ha generato varietà capostipiti, in gran parte nel Centro Italia poi diffusesi al resto della Penisola. Ruolo chiave anche del “Sangiovese” e di altre varietà-genitori. Questa ricerca ha generato un vero e proprio Atlante sulle parentele dei vitigni italiani.
Per questa ampia e diffusa ricerca, la “Parentage Atlas of Italian Grapevine Varieties as Inferred From SNP Genotyping“, sono stati coinvolti diversi centri di ricerca specializzati, enti e università. La summa dei dati raccolti in una relazione è stata pubblicata su Frontiersin.org-Frontiers in Plant Science (link).
Ecco gli enti che hanno lavorato e contribuito alla mappatura evolutiva dei vitigni italiani:
- Dipartimento di Agricoltura, Alimentazione e Ambiente, Università di Pisa, Italia;
- CREA Centro di ricerca per la genomica e la bioinformatica, Fiorenzuola d’Arda, Italia;
- CREA Centro di ricerca per la viticoltura e l’enologia, Conegliano, Italia;
- Dipartimento di Scienze della Vita, Università di Modena e Reggio Emilia, Modena, Italia;
- Dipartimento di Scienze dell’Agricoltura, Alimentazione, Risorse Naturali e Ingegneria, Università degli Studi di Foggia, Italia;
- Dipartimento di Scienze Agrarie, Alimentari e Forestali, Università degli Studi di Palermo, Italia;
- Dipartimento di Scienze Agrarie e Forestali, Università della Tuscia, Viterbo, Italia;
- Dipartimento di Scienze Agrarie, Forestali e Alimentari, Università di Torino, Italia;
- Consiglio Nazionale delle Ricerche, Istituto per la Protezione Sostenibile delle Piante, Torino, Italia.
Se ne sono occupati rispettivamente:
- Claudio D’Onofrio;
- Giorgio Tumino, Caterina Morcia, Valeria Terzi;
- Massimo Gardiman, Manna Crespan;
- Cristina Bignami;
- Laura de Palma;
- Maria Gabriella Barbagallo;
- Massimo Muganu;
- Vittorino Novello;
- Anna Schneider.
Metodo e numero di dati sui quali si è lavorato
L’intento della ricerca, oltre alla valorizzazione dei vitigni italiani, è quello di tracciare la storia delle varietà e, quindi, dei vini prodotti nel Bel Paese sin dalle epoche più lontane. Il grande lavoro è stato finanziato dal Mipaaf (progetto VIGNETO-Viticultural Characterization of the main Italian Grape Varieties and their Terroir) e dalla Fondazione AGER (progetto “An Italian Vitis database with multidisciplinary approach, for exploitation and valorization of the regional genotypes”).
Individuare i ceppi originari non è stato un compito facile e ha richiesto un lungo e duro lavoro di raffronto nei laboratori, sono stati studiati i profili genetici di centinaia di varietà che erano conservate nelle collezioni italiane ed internazionali fino a delineare un atlante delle parentele tra i vitigni italiani. Quindi, la loro evoluzione in oltre due millenni circa.
Il germoplasma dell’uva italiana è caratterizzato da un’elevata ricchezza in termini di numero di varietà, quasi 600 uve da vino iscritte nel Registro nazionale italiano delle varietà di vite e con una pletora di uve autoctone.
Nello studio è stata eseguita una genotipizzazione SNP (Single Nucleotide Polimorfisms) estesa sul germoplasma italiano di Vitis vinifera subsp (Vite selvatica) coltivata, ibridi sativa e Vitis. Diverse centinaia di varietà italiane sono poi mantenute nei depositi di istituzioni scientifiche. Sono state considerate anche circa mille varietà aggiuntive derivate da precedenti studi su Europa, Sud Italia, Magna Greciae e il germoplasma georgiano.
I grandi dati di genotipizzazione ottenuti sono stati utilizzati per verificare la presenza di omonimi e di sinonimi tra le varietà, determinare i rapporti parentali e identificare i principali antenati delle cultivar italiane tradizionali e delle adesioni strettamente correlate. È stata valutata la parentela tra un insieme di 1.232 varietà uniche e identificati un totale di 92 nuove coppie genitore-figlio (PO) e 14 nuovi trittici PO.
Materiale vegetale e preparazione del DNA
Per portare avanti le analisi e i raffronti sono state campionate complessivamente 615 campioni di vite provenienti da diverse collezioni (lista completa File 1 a questo link):
- quelle del Centro di ricerca CREA per la viticoltura e l’enologia di Conegliano (343) e dalle raccolte locali del Piemonte (79) gestite dal Consiglio Nazionale delle Ricerche e dall’Università degli Studi di Torino.
- In Emilia-Romagna e nel Lazio (54 campioni forniti) da raccolte gestite dall’Università degli Studi di Modena e Reggio Emilia e dall’Università della Tuscia.
- In Toscana (90) da patrimonio gestito dall’Università di Pisa.
- In Puglia (27 campioni) da raccolta gestita dall’Università degli Studi di Foggia;
- in Calabria e Sicilia (22 campioni aggiunti) raccolta gestita dall’Università degli Studi di Palermo.
Per l’estrazione del DNA sono stati campionati i tessuti delle foglie giovani. Il DNA genomico è stato estratto utilizzando un tampone a base di CTAB seguito dall’estrazione con cloroformio. I campioni di DNA sono stati controllati per qualità e quantità.
Le prime evidenze sulle varietà-cardine
Come sottolineato nella ricerca, la rete di parentela risultante ha suggerito che il germoplasma della vite italiana tradizionale proviene in gran parte da alcune varietà centrali geograficamente distribuite in diverse aree di influenza genetica:
- “Strinto porcino” e la sua prole “Sangiovese“, “Mantonico bianco” e “Aglianico” sono le principali varietà fondatrici dei vitigni dell’Italia sudoccidentale (IT-SW);
- Riviera Adriatica Italiana (IT-AC) e Centro Italia con la maggior parte delle varietà discendenti di “Visparola”, “Garganega” e “Bombino bianco”;
- Italia nord-occidentale (IT-NW) e Italia centrale con le principali varietà “Termarina (Sciaccarello)”, “Orsolina” e “Uva Tosca”.
È apparso chiaro il ruolo chiave di alcune cultivar e, in particolare, è apparsa chiara la centralità della “Visparola” nell’origine del germoplasma italiano. Si potrebbe ipotizzare un’ipotetica migrazione di questa varietà all’interno della Penisola Italiana da Sud a Nord lungo il versante orientale, così come del “Sangiovese” dal Sud al Centro Italia lungo il versante occidentale.
Inoltre, è stato anche evidenziato che, tra i principali fondatori di varietà di moscato, il “Moscato bianco” e lo “Zibibbo (Moscato d’Alessandria)” si sono diffusi in tutta Italia.
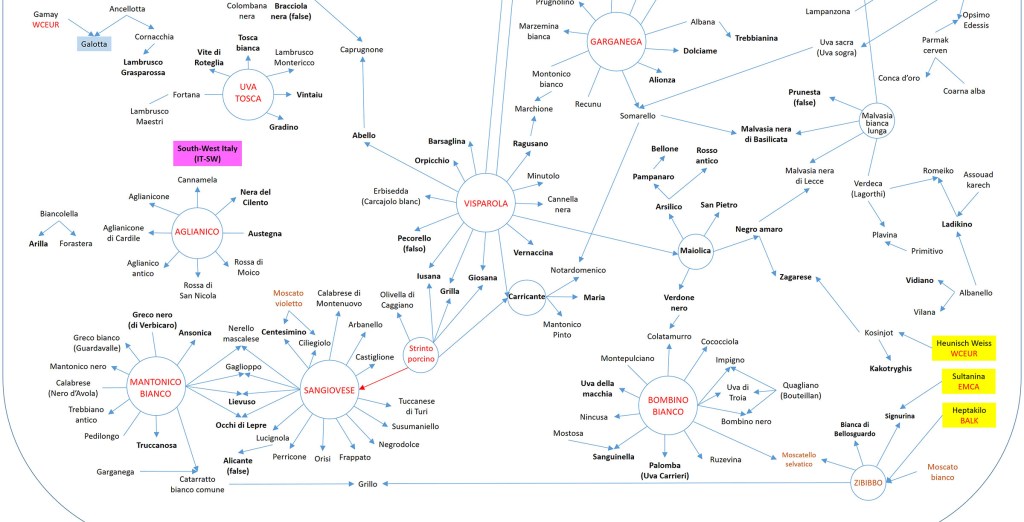
La varietà “Visparola”, considerata la sua parentela/discendenza con la varietà “Avgoustiatis” originaria del Peloponneso occidentale e delle isole greche, è risultata come genitore di tantissime altre in Italia:
- “Battraube” con “Vulpea” (originario della Pannonia, presente anche in Friuli Venezia Giulia) o SD “Baratcsuha szurke” con “Alba imputotato”;
- correlata anche al “Sangiovese” inteso come discendente dello “Strinto porcino” tipico del Sud e della Basilicata;
- e ancora, “Rossa di San Nicola” (Sud, Puglia, Calabria), “Rossa di Moico”, “Trebbiano antico”, “Palomba (Uva Carrieri) “(“Bombino bianco “), “Biancone”, “Gambuglian” (“Balzamino”), “Veltliner gruener” (“Savagnin”) e “Zemoasa” (“Heunisch Weiss”).
Inoltre, “Visparola” e “Garganega” (vitigno a bacca bianca delle province di Verona e Vicenza – DOC Soave) sono risultati nonni di “Rollo” (vitigno a bacca bianca ligure – Genova), discendente di “Termarina (Sciaccarello)” dell’Emilia-Romagna.
Gli intrecci sono molto complessi e intrecciati. “Strinto porcino” è risultato essere un genitore di “Olivella di Caggiano”.
Il “Mantonico bianco” appare apparentato a “Lucignola” e “Aglianico antico”, discendenti rispettivamente di “Sangiovese” e “Aglianico”. “Vernaccina” (figlio di “Visparola”), “Pecorino”, “Pecorello” e “Cesanese d’Affile”.
Un grande gruppo di semi-fratelli comprendeva quattro figli di “Termarina (Sciaccarello)” (“Caloria”, “Pollera nera”, “Bracciola nera” e “Rossara toscana”) insieme a “Vernaccia di Oristano (Spergola)” , “Vespaiola”, “Durella gentile” e “Albarola”.
“Famoso“, altro figlio di “Termarina (Sciaccarello)”, è risultato essere il nipote di “Alba imputotato” e di “Heunisch Weiss”.
Tanto altro ci sarebbe da scrivere, ma per i particolari più minuti basta andare direttamente alla lettura analitica del rapporto Frontiersin.org-Frontiers in Plant Science (link).






Un commento Aggiungi il tuo